前回は、画像中の領域抽出に用いられる、U-Netと呼ばれるU字型の畳み込みネットワークを紹介しました。
今回は、U-Netを人体の腹部MRI画像に適用して、実際に肝臓の領域抽出を行ってみます。
肝臓抽出に用いたU-Net
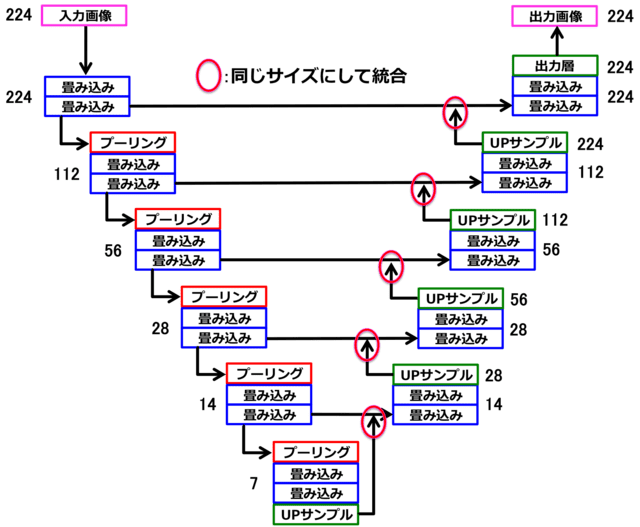
下向きパスは、「畳み込み+プーリング」により、深い層ほど特徴が局所的で位置情報が曖昧に、浅い層ほど、特徴は全体的で位置情報は正確になる。一方、上向きパスは、「畳み込み+upサンプリング」により、特徴を保持したまま、画像を大きく復元することができる。両方のパスにおいて、画像サイズが同じものを深い層から段階的にマージ(統合)することによって、局所的特徴を保持したまま全体的位置情報の復元を行うことができる。数字は、画像サイズ(pixel)を表す。
前回も解説しましたが、U-Netが領域抽出に適している理由が図1に示されています。3大特徴は、「Upサンプリング」,「Merge(マージ)」,「全結合層がない」です。
詳細は、https://lp-tech.net/articles/5MIeh (Deep learningで画像認識⑨〜Kerasで畳み込みニューラルネットワーク vol.5〜) を参照してください。
教師データの作成
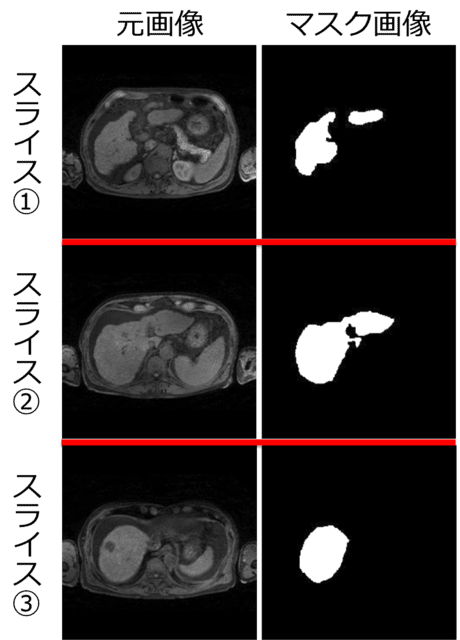
ある1人の腹部MRI画像から約10スライス毎に抜き出した7枚のスライスの内、代表的な3枚のスライス画像とそれに対応するマスク画像のセットの例。これらの組み合わせを訓練用82セット(12人)、評価用20セット(3人)作成し、オリジナルの教師データとした。その後、50倍の画像の水増しを行い、訓練用(4100スライス)と評価用(1000スライス)を追加した。水増しにおいては、元画像とマスク画像の両方に同じ移動・変形を施した。
腹部のMRI画像(3D)を用いて、領域抽出のための教師データを作成しました。
今回は、自分で肝臓領域を塗ってマスク画像を作成したので、手間を削減するために、1人の3D画像(60~70 スライス)あたり、約10スライス毎に1スライスの割合で抜き出し、計6~7 スライスのマスク画像を作成しました(画像サイズは224×224 pixel)。マスク画像の作成には、医用画像解析ソフト「3D slicer」を用いました。
訓練用に12人(82スライス)、評価用に3人(20 スライス)のMRI画像とマスク画像のセットを用意し、オリジナルの教師データとしました。図4に一例を示します。
その後、オリジナルの教師データの画像データセット数を50倍水増し(Data Augmentation)し、訓練用(4100スライス)と評価用(1000スライス)を追加しました。当然ですが、Data Augmentationにおいて、元画像とマスク画像の両方に同じ移動・変形を施す必要があります。
Kerasでは、Data Augmentationにおける関数(Image Generator)が既に提供されていますが、関数を自作することもできます。また、学習の繰り返し計算の中で水増しすることもできますが、もちろん前もって水増ししておくこともできます。今回は、自作の関数を用いて、前もって水増しを行いました。医用画像の場合、あまりにも不自然な移動・変形を加えて水増しすることは得策ではない気がします。例えば、画像を左右反転させて肝臓が左側になる、というような変形は行うべきではないでしょう。
推測結果
領域抽出では、評価値としてDice(ダイス)係数というものを使います。教師データであるマスク画像と推測領域との類似度を示す指標です。下のように、通常のCNNでaccuracy, val_accuracyの箇所が、dice_coef, val_dice_coefになっているのが分かります。
また、出力層のシグモイド関数の閾値は0.5として白黒画像に変換しました。
Epoch 92/1000
4182/4182 [==============================] - 99s - loss: -0.8884 - dice_coef: 0.8884 - val_loss: -0.8273 - val_dice_coef: 0.8273
Epoch 93/1000
4182/4182 [==============================] - 99s - loss: -0.8887 - dice_coef: 0.8887 - val_loss: -0.8197 - val_dice_coef: 0.8197
Epoch 94/1000
4182/4182 [==============================] - 99s - loss: -0.8885 - dice_coef: 0.8885 - val_loss: -0.8297 - val_dice_coef: 0.8297
results.py最適化ループの回数(epoch数)を増やすと訓練画像の評価値は徐々に増えて行きますが、評価画像の評価値は、100回程度のepoch数で約0.83に収束しました(図2)。
そこで、epoch数100回付近で評価画像の評価値が最大となるモデル重みを用いて、テスト画像(ある1人のMRI画像)から肝臓領域の推測を行いました(図6)。推測領域に若干欠如が見られますが、訓練データの情報量が元の3Dデータの約1/10(約10スライス毎に抜き出して作成したため)である割には、かなり高い精度で推測が行われたと考えられます。

評価値は、推測領域とマスク画像との類似度を示すDice係数(縦軸)。最適化ループの回数(epoch数)を増やすにつれ訓練画像の評価値は徐々に増え0.9を越えて行くが、評価画像の評価値は、100回程度のepoch数で約0.83に収束した。
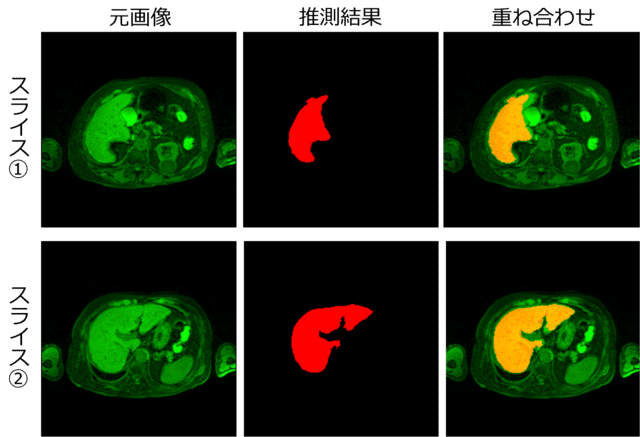
評価画像の評価値が最大となるモデル重みを用いて、テスト画像(ある1人のMRI画像)から肝臓領域の推測を行った。代表的な2枚のスライスにおける結果を示す。元画像(左:緑色)、推測領域(中央:赤色)。右は両者を重ね合わせた結果で、重複領域が黄色で表示されている。
今回は、このU-Netを用いて、実際のMRI画像から肝臓領域を抽出してみました。
訓練データの情報量が元の3Dデータの約1/10である割には、かなり高い精度で推測が行われたと考えられます。
このように、U-netによってかなり精度良く臓器抽出を行うことが可能です。
手持ちのデータがある方は、是非色々試してみてはいかがでしょうか?
【参考文献 / URL】
[1] https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/
[2] Fully Convolutional Networks for Semantic Segmentation, Evan Shelhamer, Jonathan Long, and Trevor Darrell, Member, IEEE, 2016.
[3] 実装ディープラーニング,藤田一弥, 高原 歩,オーム社.