前回までは、畳み込みネットワークを用いて画像に「何が」写っているかの推測(クラス分類)を行いましたが、今回は、U-Netと呼ばれるU字型の畳み込みネットワークを用いて、画像中に「何が、どこに、どのように」写っているのか(領域抽出)を行います。
人体の腹部MRI画像にU-Netを適用して、肝臓の領域抽出を行ってみます(今回は前準備です)。
ディープラーニングを用いた物体検出・領域抽出
畳み込みニューラルネット(CNN)は、画像のクラス分類(画像認識)だけでなく、物体の位置や大きさの検出(物体検出)や形状の抽出(領域抽出)にも用いられます(図1, 2)。例えば、物体検出には、R-CNN, Fast R-CNN, Yoloなどが、領域抽出にはSegNetやU-Netなどのニューラルネットが用いられます。

物体検出には、R-CNN, Fast R-CNN, Yoloなどのニューラルネットワークが用いられる。特にYoloは、その他の100~1000倍高速に検出できるため、動画に対する物体検出に用いられる。
via pjreddie.com
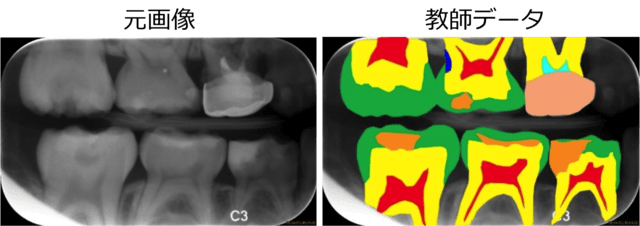
歯のX線写真から虫歯を探すコンテスト(ISBI 2015)で用いられた画像。
(左)元画像, (右)領域ごとに塗り分けられた教師データ。これらをU-Net(後述)に学習させ、物体の位置と形状の推測(領域抽出)を行う。
via www-o.ntust.edu.tw
U-Net(U字型のニューラルネットワーク)
通常のCNNによって行われる画像のクラス分類(画像認識)では、畳み込み層が物体の局所的な特徴を抽出する役割を担い、プーリング層が物体の全体的な位置情報をぼかす(位置ズレの許容)の役割を担っています。そのため、より深い層ほど、抽出される特徴はより局所的になり、その特徴の全体的な位置情報はより曖昧になります。言い換えれば、プーリング層のおかげで、物体の位置ズレや大きさの違いの影響をあまり受けない、頑強なパターン認識が可能になっているわけです。
一方、領域抽出では、「物体の局所的特徴と全体的位置情報」の両方を元画像上で特定しなければなりません。つまり、プーリング層でぼかされた局所的特徴の位置情報を元画像上でpixel単位で正確に復元する必要があります。
そこで、「物体の局所的特徴と全体的位置情報」の両方を統合して学習させるために開発されたのがU-Netです[1]。図3に示すように、U字型のネットワークになっていることから名付けられました。
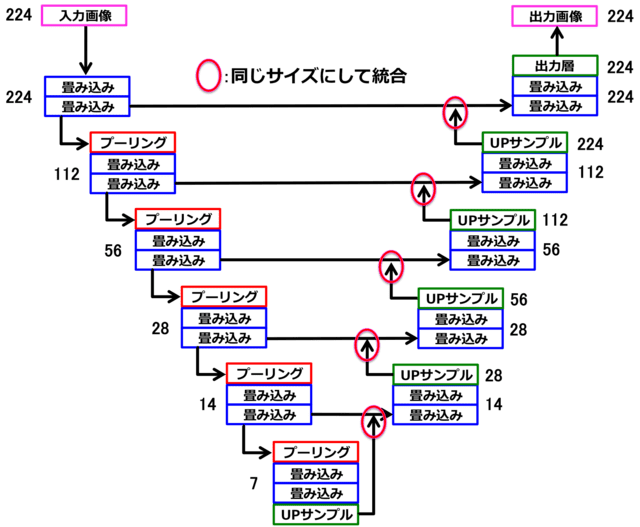
下向きパスは、「畳み込み+プーリング」により、深い層ほど特徴が局所的で位置情報が曖昧に、浅い層ほど、特徴は全体的で位置情報は正確になる。一方、上向きパスは、「畳み込み+upサンプリング」により、特徴を保持したまま、画像を大きく復元することができる。両方のパスにおいて、画像サイズが同じものを深い層から段階的に統合することによって、局所的特徴を保持したまま全体的位置情報の復元を行うことができる。数字は、画像サイズ(pixel)を表す。
U-Netが通常のCNNと異なる3大特徴は、
「Upサンプリング」,「Merge(マージ)」,「全結合層がない」です。
① Upサンプリング
上向きパスにおける「upサンプリング」と呼ばれる操作で、次元を上げる(画像を大きく復元する)意味があります。次元を下げる(画像を小さく圧縮する)プーリングの逆の効果を持つため「アンプーリング」とも呼ばれます。
② Merge(マージ)
文字通り情報を統合する意味があります。U-Netの下向きパスは、深い層ほど、特徴が局所的で位置情報が曖昧に、浅い層ほど、特徴は全体的で位置情報は正確になります。上向きパスは、特徴を保持したまま、画像を大きく復元することができるので、両方のパスにおいて、画像サイズが同じものを深い層から段階的にマージすることによって、局所的特徴を保持したまま全体的位置情報の復元を行うことができます。
マージにも色々な種類がありますが、今回のU-Netでは、「チャンネルとして追加する」という意味でマージを行います。例えば、図3の一番深い層におけるマージは、Kerasを使うと下のように表されます。5段目の畳み込み後の出力(conv5)と6段目の畳み込み後の出力(conv6)をupサンプリング(2×2)したものをconcat型で(チャンネルとして)結合させる、という意味です。
up7 = merge([UpSampling2D(size=(2, 2))(conv6), conv5], mode='concat', concat_axis=3)
merge.py③ 全結合層がない
通常のCNNに見られるような全結合層がありません。クラス分類では、全結合層が必要ですが、領域抽出では画像が出力になるため、全結合層が必要ありません。そのため、畳み込み層だけのネットワーク = Fully Convolutional Network(FCN)と呼ばれます[2]。出力層では、シグモイド関数を使用することにより、出力は0~1の値となるため、閾値を設けて、白黒の二値画像として出力させます。
今回は、領域抽出に用いられるU-Netを紹介しました。
次回、このU-Netを用いて、実際のMRI画像から肝臓領域を抽出してみます。
U-Netの構造部分をKerasで書いたものを下に載せておきますので、参考にしてください(但し、バックエンドはTensorflow)。
【参考文献 / URL】
[1] https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/
[2] Fully Convolutional Networks for Semantic Segmentation, Evan Shelhamer, Jonathan Long, and Trevor Darrell, Member, IEEE, 2016.
[3] 実装ディープラーニング,藤田一弥, 高原 歩,オーム社.
def create_fcn(input_size):
inputs = Input((input_size[1], input_size[0], 3))
conv1 = Convolution2D(32, 3, 3, activation='relu', border_mode='same')(inputs)
conv1 = Convolution2D(32, 3, 3, activation='relu', border_mode='same')(conv1)
pool1 = MaxPooling2D(pool_size=(2, 2))(conv1)
conv2 = Convolution2D(64, 3, 3, activation='relu', border_mode='same')(pool1)
conv2 = Convolution2D(64, 3, 3, activation='relu', border_mode='same')(conv2)
pool2 = MaxPooling2D(pool_size=(2, 2))(conv2)
conv3 = Convolution2D(128, 3, 3, activation='relu', border_mode='same')(pool2)
conv3 = Convolution2D(128, 3, 3, activation='relu', border_mode='same')(conv3)
pool3 = MaxPooling2D(pool_size=(2, 2))(conv3)
conv4 = Convolution2D(256, 3, 3, activation='relu', border_mode='same')(pool3)
conv4 = Convolution2D(256, 3, 3, activation='relu', border_mode='same')(conv4)
pool4 = MaxPooling2D(pool_size=(2, 2))(conv4)
conv5 = Convolution2D(512, 3, 3, activation='relu', border_mode='same')(pool4)
conv5 = Convolution2D(512, 3, 3, activation='relu', border_mode='same')(conv5)
pool5 = MaxPooling2D(pool_size=(2, 2))(conv5)
conv6 = Convolution2D(1024, 3, 3, activation='relu', border_mode='same')(pool5)
conv6 = Convolution2D(1024, 3, 3, activation='relu', border_mode='same')(conv6)
up7 = merge([UpSampling2D(size=(2, 2))(conv6), conv5], mode='concat', concat_axis=3)
conv7 = Convolution2D(512, 3, 3, activation='relu', border_mode='same')(up7)
conv7 = Convolution2D(512, 3, 3, activation='relu', border_mode='same')(conv7)
up8 = merge([UpSampling2D(size=(2, 2))(conv7), conv4], mode='concat', concat_axis=3)
conv8 = Convolution2D(256, 3, 3, activation='relu', border_mode='same')(up8)
conv8 = Convolution2D(256, 3, 3, activation='relu', border_mode='same')(conv8)
up9 = merge([UpSampling2D(size=(2, 2))(conv8), conv3], mode='concat', concat_axis=3)
conv9 = Convolution2D(128, 3, 3, activation='relu', border_mode='same')(up9)
conv9 = Convolution2D(128, 3, 3, activation='relu', border_mode='same')(conv9)
up10 = merge([UpSampling2D(size=(2, 2))(conv9), conv2], mode='concat', concat_axis=3)
conv10 = Convolution2D(64, 3, 3, activation='relu', border_mode='same')(up10)
conv10 = Convolution2D(64, 3, 3, activation='relu', border_mode='same')(conv10)
up11 = merge([UpSampling2D(size=(2, 2))(conv10), conv1], mode='concat', concat_axis=3)
conv11 = Convolution2D(32, 3, 3, activation='relu', border_mode='same')(up11)
conv11 = Convolution2D(32, 3, 3, activation='relu', border_mode='same')(conv11)
conv12 = Convolution2D(1, 1, 1, activation='sigmoid')(conv11)
fcn = Model(input=inputs, output=conv12)
return fcn
U-Net.py